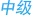IDT NGS二代测序文库均一化试剂盒
--获得高度稳定且均一化处理的DNA/RNA测序文库
北京云肽生物科技代理的美国进口IDT xGen Normalase 文库均一化试剂盒,采用了一种新型酶促文库均一化处理技术,可对|||umina测序仪测序前的DNA或RNA文库进行均一化处理。通过IDT文库均一化试剂盒,上机前无需进行单个文库浓度检测,可得到优化的簇密度和更均衡的文库。IDT文库均一化处理试剂盒可搭配常规建库实验流程,不仅能缩短实验整体的操作时间,还能提高NGS文库上机浓度的精确性,实现混合文库间测序数据量的均一性。IDT文库均一化处理实验流程包括文库的非偏好件选择、酶均一化反应,在文库扩增中使用带有Normalase标记的引物,可稳定得到3倍于目标均一化浓度的产量。例如上机前文库浓度需要均一化为2nM或4nM的标准文库产量,则IDT均一化反应前文库浓度至少分别为6nM或12M(20ul体积)。该实验过程中不需要增加额外PCR步骤,仅需将常规文库扩增引物替换成带有Normalase标记的测序通用引物或带有样本标签的引物。IDTxGen Normalase 文库均一化试剂盒为高通量实验室提供了快速、可扩展的文库标准化工作流程。
优势
①节省时间,提高通量:统一的样本处理过程,生成平衡均一的混合文库;
②减少文库测序差异,降低测序费用:得到更平衡的混合文库,允许更多的样本混合上机,降低测序成本;
③适用多种工作流程,设计灵活:兼容多种文库制备方法,获得均一平衡的测序数据。
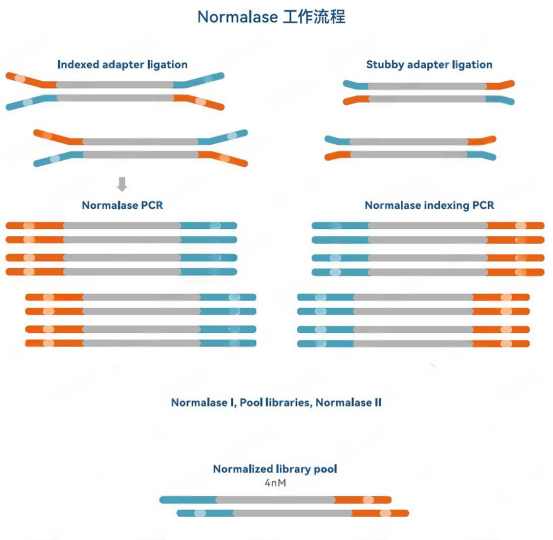 |
图.IDT xGen Normalase 文库均一化试剂盒的工作流程始于建库接头连接反应后,工作流程根据使用全长或短接头而不同。通过NormalasePCR,将文库扩增到均一化处理反应所需zui低数量,然后进行下游的酶均一化处理。在此过程中,如连按反应中使用全长接头,PCR扩增需使用带有Nomalase标记的测序通用引物;如使用非全长接头,则需使用带有Normalase标记的样本标签作为扩增引物。将扩增好的单个NGS文库分别进行15分钟Normalasel孵育,此反应过程使用酶促法,可以无偏好性地选择指定摩尔浓度的NGS文库。之后,将每个文库等体积的混合到一管中,进行15分钟Normalase ll孵育,酶促法将每个NGS文库标准化到指定的摩尔浓度。实现上机测序前,得到平衡且均一化的NGS混合文库。
一、
 |
不同片段大小,也可得到高度稳定且均一化测序文库
表.使用lDT Normalase试剂盒将文库均一化处理成4nM,上样浓度为12pM,得到和预期一致的簇密度(lllumina MiSeq测序,V2版本试剂)。
二、相比传统上机定量方式,可得到数据量更均一的混合文库
 |
图、测试分别来自于两名实验者(n=16/每名实验者)的32个样品,使用IDT xGen DNA Library PrepEZ 试剂盒构建文库。实验样本为NA12878 gDNA,起始量为1~250ng。NormalasePCR后的文库使用qPCR方法定量,保证文库达到Normalase反应所需zui低阈值。"qPCR":基于qPCR定量结果,对文库进行均一化处理、混样和测序;"Normalase":同样样品使用Normalase试剂盒,对文库进行混样、均一化处理和测序。比较两种方法每种接头测序产生数据百分比(lllumina Miseq v2的50循环测序试剂盒)。结果显示:qPCR混含文库中文库测序数量变异系数(CV)为22.5%,而Normalase混合文库池的变异系数为9.4%(中位线为95%的置信区间)。
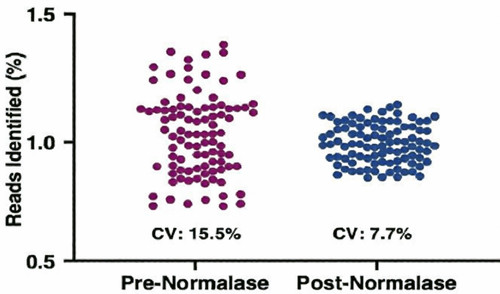 |
图.10ngNA12878 gDNA,使用IDT xGen DNA Library Prep EZ试剂盒构建96个文库,搭配IDT xGen CDl接头引物进行扩增。文库混样、均一化处理前,使用Qubit定量结果做等体积的文库混样,之后使用lllumina Miseq V2测序试剂盒上机测序(50个循环),通过每种接头产生测序数据量百分比进行比较。均一化反应之前混合文库池CV为15.5%,证明Normalase引物的扩增结果稳定、重复性好。将相同的文库均一化处理到4nM,并使用lllumina Miseq V250循环测序试剂盒上机测序。均一化处理后,文库CV降为7.7%,证明通过IDT xGen Normalase可得到更加均一化的混合文库(中位线为95%置信区间)。
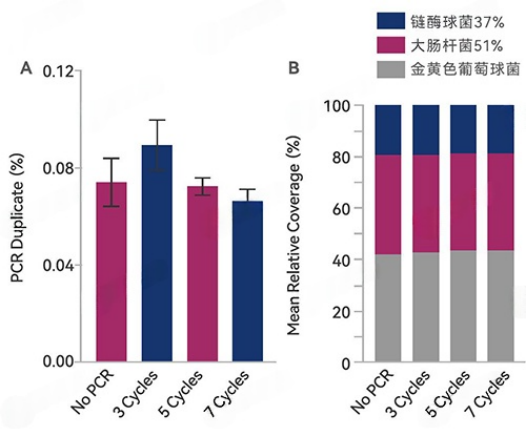
图.针对不同GC%含量基因组细菌的测序覆盖度及冗余率结果
使用IDT xGen DNALibrary Prep EZ建库试剂盒,起始量为100ng DNA,包含三种不同比例混合的參考菌株(大肠杆菌、金黄色葡萄球菌、链酶球菌)。片段化DNA至350bp,建库时搭配全长的Y型接头进行连接,使用Normalase标记的通用测序引物扩增3、5或7个循环。依据qPCR定量结果进行文库混合,并使用lllumina MiSeq PE150测序。测序结果显示,不同GC%含量基因组细菌的测序覆盖度及冗余率没有显著差异。
 |
图.使用IDT文库均一化试剂盒,对插入片段大小即菌种覆盖度评估无影响
将5ng的MSA-1000样品(GC%基因组含量不同的10种等比例混合菌株),用IDT xGen DNA Lilbrary PrepEZ 试剂盒构建文库,分别用IDT Normalase标记引物(紫红色)和IDT普通引物(蓝色)对两个文库进行扩增。使用IDT均一化试剂盒或Qubit将文库均一化处理为4nM。进行lllumina MiniSeq 平台高通量上机(PE-150)测序。实验结果显示,所构建的基因组文库中(由不同GC%含量的细菌组成),经IDT均一化处理的文库,仍保持养稳定插入片段大小(A)和高基因组覆盖度(B)。
三、文库均一化试剂盒的使用,不影响原有RNA测序数据分析结果
平行对比RNA文库进行上机前均一化处理使用IDT文库均一化试剂盒(n=2)、GPCR方法(n=2)的效果。4个RNA-seq文库(50ng人脑mRNA样本)分别使用带有Normalase标记的双端样本标签、常规双端样本标签进行文库制备。将带有常规样本标签的文库(标记为"1"和"2"号文
 |
库),按qPCR定量结果进行文库均一化及混样。将带有Nomalase标记的文库(标记为"3"和“4号文库),在文库制备完成后,搭配IDT文库均一化试剂盒,将RNA文库均一化处理至4nM,通过Miniseq(2x150)上机测序,证明IDT文库均一化试剂盒对RNA-Seq分析结果无影响。将各文库均一化处理至4nM后上机测序,使用STAR、Picard及RSeQC进行比对、进行RNA·sec数据分析。韦恩图结果显示,每个样本中检测到的qian一千个转录本结果没有显著差异。
表.相同RNA文库,分别使用qPCR文库定量、IDT Normalase文库均一化试剂盒进行文库均一化处理,其RNA·seq数据分析结果比较。由于IDT试剂盒使用非偏好性的酶促方法进行文库均一化处理,因而两种文库均一化处理有着近似的RNA-seq分析结果,与预期相符。
 |
表.相同RNA文库,分别使用qPCR文库定量、IDT Normalase文库均一化试剂盒进行文库均一化处理,其1000个高表达转录本分析结果比较。由于IDT试剂盒使用非偏好性的酶促方法进行文库均一化处理,因而二者的1000个高表达转录本RNA-seq分析结果近似,与预期相符。

图.韦恩图显示每个样本中检测到的qian 一千个转录本结果没有显著差异

IDT文库均一化试剂盒优势
①可灵活调整均一化处理后文库浓度,只需样本扩增后浓度超过最少文库量阈值,即可保证最终所需文库浓度均一化;
②处理后的均一化文库浓度为4nM,选择使用≥6nM均一化工作流程时,最终文库浓度为2nM;
③混合文库间测序变异系数系数≤10%;
④试剂盒兼容性强,可兼容全长测序接头或短接头的建库流程;兼容非靶向富集的工作流程,制备的文库可直接测序(如全基因组测序或全转录组测序);兼容杂交捕获流程,即靶向富集完成后上机前的文库均一化。需保证均一化反应前,有稳定的文库产量(20μL反应体系中文库摩尔浓度≥6nM或≥12nM),以得到标准的2nM/4nM文库量;
⑤兼容IDT xGen接头、IDT xGen Normalase CDl引物、IDT xGen Normalase UDl引物;
⑥兼容IIlumina测序平台,并可获得一致的测序结果。
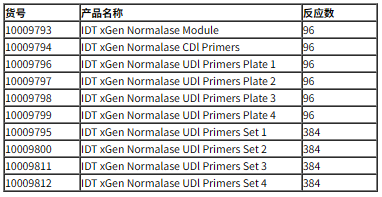
北京云肽生物科技代理的IDT文库均一化试剂盒产品
 |